NGS(Next Generation Sequencing)란 무엇일까?
NGS 검사 또는 유전자 검사라는 단어를 많이 들어보셨을까요?
일반적으로 유전자 검사라고 하면 친자확인을 많이 떠올리실 것 같습니다.
하지만, 최근 병원에서도 질병을 진단하거나 개인에게 맞는 치료제 또는 항암제를 찾거나 하는 등 다양한 목적으로 유전자 검사가 진행되고 있습니다.
유전자 검사라 함은 개인이 본래 가지고 있는 유전자의 염기 서열을 읽음으로써 유전적으로 돌연변이가 있는지, 비만 발생 유전자를 가지고 있는지, 항생제 내성을 가지고 있는지, 어떠한 질병에 대한 위험도를 가지고 있는지 미리 예측해 볼 수 있는 등 본인의 유전자에 대한 특성을 파악하는 방법입니다.
이번 포스트에서는 본인의 유전자 염기 서열을 읽는 방식 중 하나이며, 최근 많이 사용화되어있는 방법인 NGS라는 것이 무엇인지 알아보도록 하겠습니다.
1. NGS의 개요
기존의 직접염기서열분석법(direct sequenicng)은 분석하고자 하는 부위를 PCR 증폭해야 하기 때문에 여러 타겟을 분석할 경우 많은 시간과 노력 및 비용이 소요되어 효율성이 낮은 문제점이 있었습니다. 이러한 단점을 극복하고자 차세대 염기서열분석(next generation sequencing; NGS) 법이 개발되었으며 이것은 DNA 가닥을 각각 하나씩 분석하는 방식으로 기존의 직접 염기서열분석법에 비해 매우 빠르고 저렴하게 염기서열이 가능하다는 장점을 가지고 있습니다. NGS는 DNA를 일정한 조각(fragment)으로 분절화시키고 장비가 인식할 수 있는 특정 염기서열을 가진 올리고뉴클레오티드(oligonucleotide)를 붙여주는 라이브러리(library) 제작, 각 라이브러리 DNA 가닥의 염기서열을 장비에서 읽는 단계, 그리고 장비에서 생성된 데이터를 가공하여 알고리즘으로 분석하는 단계로 구성됩니다.


2. NGS 장비의 종류
NGS를 할 수 있는 장비는 현재 여러 군데의 회사에서 각 회사만의 기술 및 다른 원리를 바탕으로 만들어져 출시되어 있습니다.
전세계적으로 가장 많이 쓰이는 NGS 장비인 현재 일루미나(Illumina)와 그 외에 써모피셔사이언티픽(Thermo Fisher Scientific), 퍼시픽바이오사이언스(Pacific Biosciences), 옥스포드나노포어(Oxford Nanopore Technologies) 등의 회사에서 출시된 제품들이 관련 시장을 형성하고 있습니다. 퍼시픽바이오사이언스, 옥스포드나노포어 등의 3세대 장비들은 2세대 장비의 짧은분석범위의 단점을 보완한 것으로서 PCR 증폭 과정이 없이 DNA 단일분자를 실시간(real-time)으로 읽어매우 긴 DNA 가닥의 염기서열을 한번에 읽을 수 있다는 장점이 있으나 개별 염기서열 측면에서 보면정확도가 높지 않은 단점이 있습니다.
그렇다면 각 회사의 장비가 어떠한 원리를 바탕으로 NGS가 수행되어 지며, 어떤 특징을 가지고 있는지 간단하게 알아보도록 하겠습니다.
1) 일루미나Illumina 장비
일루미나(Illumina) 사의 기술은 DNA 가닥을 복제 혹은 합성하면서 각기 다른 형광이 부착된 염기(dATP, dCTP, dTTP, dGTP)와 함께 DNA 중합효소(polymerase)를 넣어 주면 각 DNA의 염기서열에 상보적인 염기가 삽입되면서 각기 다른 형광을 방출하게 되는데 이것을 장비에 부착된 CCD 카메라로 사진을 찍고, 장비는 형광이 발생된 클러스터의 위치를 기억하고 있다가 각 클러스터의 형광 변화를 감지하여 클러스터의 염기서열을 기록합니다. 출시된 장비로는 iSeq, MiniSeq, MiSeq, NextSeq, HiSeq, NovaSeq 등이 있으며열거된 순서에 따라 출력(output)이 높아져 대량의 데이터를 생산할 수 있습니다.

2) 써모피셔사이언티픽(Thermo Fisher Scientific) 장비
써모피셔사이언티픽(Thermo Fisher Scientific) 사의 장비는 인수하기 전 처음 개발한 회사인 Ion Torrent 장비로 더 널리 알려져 있습니다. 이 장비는 미세한 비드(bead)에 DNA 라이브러리를 붙여 촘촘하게 짜여진 미세한 구멍(well)에 하나씩 들어가도록 하고 각 구멍 밑에는 전류를 측정할 수 있는 반도체 칩 회로가 설계되어 있어 상보적인 염기가 결합하는 합성 과정에서 방출되는 수소이온(H+)으로 인하여 pH가 낮아지는 것을 반도체칩의 센서가 탐지하고 분석하는 방식입니다. 현재 출시된 장비로는 Ion Personal Genome Machine(PGM) 및 Ion S5 System 두 가지 종류가 있습니다.

3. NGS 전처리 과정
NGS 전처리 과정은 샘플 DNA 혹은 RNA가 NGS 장비에서 인식하여 염기서열분석을 할 수 있도록 가공하는 과정입니다. NGS 장비가 인식하기 위해서는 보통 샘플 DNA 혹은 RNA를 적당한 크기로 자른 후 양 끝에 특정한 염기서열을 가진 올리고뉴클레오티드(oligonucleotide)를 붙여준다. 이렇게 만들어진 산물을 라이브러리(library)라고 부릅니다.

1) 라이브러리 제작 - Library Preparation
라이브러리 제작은 분절화(fragmentation), 말단수리(end-repair), 3′-말단 아데닌 추가(A-tailing), 어댑터 부착(adapter ligation), 라이브러리 증폭(library amplification)으로 나누어집니다. 샘플에서 추출된 핵산은 NGS 장비에서 분석 가능한 크기로 적절하게 절단시켜야 합니다. 이것을 분절화(fragmentation)라 부르고 이은 물리적 또는 효소적인 방법으로 무작위로 절단을 일으킵니다. 분절화가 끝난 핵산은 불완전한 말단을 수선해 주고(end-repair) 3′-끝에 아데닌(adenine) 염기를 추가하여 어댑터 부착이 쉽게 만듭니다. 어댑터(Adapter)란 NGS 장비가 인식할 수 있는 고유한 염기서열을 가진 올리고뉴클레오티드로서, DNA에 어댑터를 붙여 주는 작업은 보통 리게이즈(ligase) 효소를 이용합니다. 어댑터가 붙은 라이브러리는 NGS 검사를 하기에 양이 충분하지 않기 때문에 PCR 증폭 과정을 거치고 마지막으로 예기치 않은 부산물 등을 제거하는 정제(clean-up) 과정을 거칩니다.
2) 타겟 선별 - Target enrichment
만들어진 라이브러리를 추가 작업 없이 NGS 분석을 하여 모든 DNA의 데이터를 얻는 것을 whole genome sequencing(WGS)이라 하고, 모든 RNA의 정보를 분석하면 whole RNA sequencing이 됩니다. 원하는 유전자 부위만 보고자 한다면 분석하고자 하는 부분의 DNA 혹은 RNA를 선별해야 하며 이것을 타겟 선별(target enrichment)이라 하고 이렇게 NGS 분석을 하는 것을 타겟 패널 시퀀싱(targeted sequencing)이라 부릅니다. 타겟 선별은 PCR 프라이머(primer)로 증폭을 하는 앰플리콘(amplicon) 방법과 프로브(probe)를 이용하여 교합(hybridization)하는 캡쳐(capture) 방법으로 나뉩니다. PCR 앰플리콘 방식은 검사 소요시간이 더 짧고, 상대적으로 적은 양의 DNA를 필요로 하여 잘 디자인된 작은 수의 유전자 패널에 대한 검사에 유용하지만, 패널의 유전자 수가 많아지거나 엑솜 시퀀싱(exome sequencing)을 수행하는 경우에는 프로브 방식이 유리합니다.
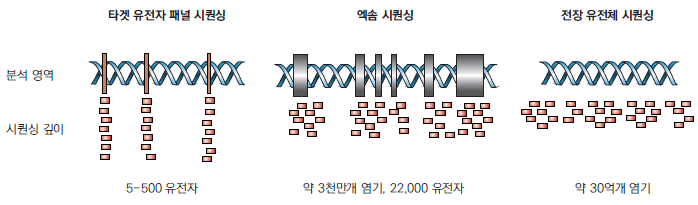
A. 전장 유전체 시퀀싱 - Whole genome sequencing, WGS
유전체 전체를 분석하는 방법으로서 타겟 선별 단계가 필요 없으나 인트론(intron)을 포함한 광범위한 영역을 분석하므로 시퀀싱 비용은 크게 증가하며 상대적으로 각 영역별 시퀀싱 깊이(depth)는 낮아지기 때문에 분석정확도가 낮아집니다. 대신 인트론과 비번역 부위(untranslated region)을 분석할 수 있기 때문에 구조적(structural) 변이나 유전자 발현 조절과 관련된(regulatory) 변이를 검출할 수 있는 장점이 있습니다.
B. 엑솜 시퀀싱 - Whole exome sequencing, WES
인간의 유전자는 약 22,000개 이상이 존재하며 엑손(exon) 부위는 단백질을 직접 코딩하는 부위로 전체 유전체의 유전체의 1~2% 정도를 차지합니다. 대부분의 질환 연관 돌연변이는 이 위치에 존재하므로 엑솜 시퀀싱으로 효과적으로 돌연변이를 검출할 수 있습니다. 엑솜 시퀀싱은 중간 정도의 시퀀싱 깊이를 얻을 수 있으며 전장 유전체 시퀀싱에 비하여 비용이 저렴하고 분석에 소요되는 시간이 줄어들어 효율적입니다.
C. 타겟 패널 시퀀싱 - Targeted panel sequencing
타겟 패널 시퀀싱은 특정 질병이나 증상의 원인이 되는 유전자들로만 구성된 패널을 구성하여 검사하는 방법으로서 하나의 질환과 관련된 유전자가 여러 개인 경우 유용합니다. 이 방법은 몇 개의 유전자를 선택적으로 검사하므로, 엑솜 시퀀싱이나 전장 유전체 시퀀싱에 비하여 높은 시퀀싱 깊이(depth)를 얻을 수 있어 정확도가 높고 비용이 저렴하기 때문에 현재 임상 검사로 가장 많이 사용되고 있습니다.
기존의 직접염기서열분석법(direct sequenicng)은 분석하고자 하는 부위를 PCR 증폭해야 하기 때문에 여러 타겟을 분석할 경우 많은 시간과 노력 및 비용이 소요되어 효율성이 낮은 문제점이 있었습니다. 이러한 단점을 극복하고자 차세대 염기서열분석(next generation sequencing; NGS) 법이 개발되었으며 이것은 DNA 가닥을 각각 하나씩 분석하는 방식으로 기존의 직접 염기서열분석법에 비해 매우 빠르고 저렴하게 염기서열이 가능하다는 장점을 가지고 있습니다. NGS는 DNA를 일정한 조각(fragment)으로 분절화시키고 장비가 인식할 수 있는 특정 염기서열을 가진 올리고뉴클레오티드(oligonucleotide)를 붙여주는 라이브러리(library) 제작, 각 라이브러리 DNA 가닥의 염기서열을 장비에서 읽는 단계, 그리고 장비에서 생성된 데이터를 가공하여 알고리즘으로 분석하는 단계로 구성됩니다.


이상으로 NGS에 대해서 대략적으로 알아보았습니다. 다음 포스팅에 이어서 NGS에 대해 좀 더 작성해보도록 하겠습니다.
출처: 식품의약품안전평가원 식품의약품안전처 - 차세대염기서열분석 해설서