가. 전장 유전체 시퀀싱 - Whole genome sequencing, WGS
전장 유전체 시퀀싱은 유전체 전체를 분석하는 방법으로서 타겟 선별 단계가 필요 없으나 인트론(intron)을 포함한 광범위한 영역을 분석하므로 시퀀싱 비용은 크게 증가하며 상대적으로 각 영역별 시퀀싱 깊이(depth)는 낮아지기 때문에 분석정확도가 낮아진다. 대신 인트론과 비번역 부위(untranslated region)을 분석할 수 있기 때문에 구조적(structural) 변이나 유전자 발현 조절과 관련된(regulatory) 변이를 검출할 수 있는 장점이 있다.
나. 엑솜 시퀀싱 - Whole exome sequencing, WES
인간의 유전자는 약 22,000개 이상이 존재하며 엑손(exon) 부위는 단백질을 직접 코딩하는 부위로 전체 유전체의 1~2% 정도를 차지한다. 대부분의 질환 연관 돌연변이는 이 위치에 존재하므로 엑솜 시퀀싱으로 효과적으로 돌연변이를 검출할 수 있다. 엑솜 시퀀싱은 중간 정도의 시퀀싱 깊이를 얻을 수 있으며 전장 유전체 시퀀싱에 비하여 비용이 저렴하고 분석에 소요되는 시간이 줄어들어 효율적이다.
다. 타겟 패널 시퀀싱 - Targeted panel sequencing
타겟 패널 시퀀싱은 특정 질병이나 증상의 원인이 되는 유전자들로만 구성된 패널을 구성하여 검사하는 방법으로서 하나의 질환과 관련된 유전자가 여러 개인 경우 유용하다. 이 방법은 몇 개의 유전자를 선택적으로 검사하므로, 엑솜 시퀀싱이나 전장 유전체 시퀀싱에 비하여 높은 시퀀싱 깊이(depth)를 얻을 수 있어 정확도가 높고 비용이 저렴하기 때문에 현재 임상 검사로 가장 많이 사용되고 있다.


| 1 | PCR 엠플리콘(amplicon) 방식
1) 표적 중합효소연쇄반응 - Targeted PCR
중합효소연쇄반응(PCR) 방식의 타겟 선별은 일반적인 forward 및 reverse 프라이머(Primer)를 제작하여 원하는 유전자의 부위를 증폭시킨다. 하나의 PCR 튜브에 한 세트의 프라이머를 사용하여 증폭하는(singleplex PCR) 일반적인 방법도 가능하고 하나의 PCR 튜브에 여러 세트의 프라이머를 한 번에 넣어 주어 다양한 표적을 한꺼번에 검출할 수 있는 다중중합효소 연쇄반응(multiplex PCR)도 사용될 수 있다. 다중 중합효소 연쇄반응은 프라이머 사이의 간섭으로 인해 많은 유전자 부위들을 균일하게 증폭하는 것이 까다롭지만 실험이 한 튜브에서 진행되기 때문에 적은 양의 시료에서 다양한 유전자 부위를 적은 비용으로 검출할 수 있는 장점이 있다.

2) 어댑터 부착 및 인덱스 PCR - Adapter ligation and index PCR
NGS 장비에서 인식할 수 있는 어댑터를 부착하기 위해서는 PCR 증폭 산물을 리게이즈(ligase)를 이용할 수 있다. 혹은 PCR 프라이머에 어댑터 염기서열 일부를 포함하도록 고안하여 1차 PCR 증폭을 진행한 후 1차 PCR 프라이머 끝부분을 포함한 어댑터 프라이머를 이용하여 2차 PCR 증폭을 하면 완전한 어댑터가 포함된 라이브러리를 만들 수 있으며 이것을 fusion PCR 법이라고도 부른다. 2차 PCR 프라이머에는 샘플 인덱스를 포함하도록 디자인할 수 있다.
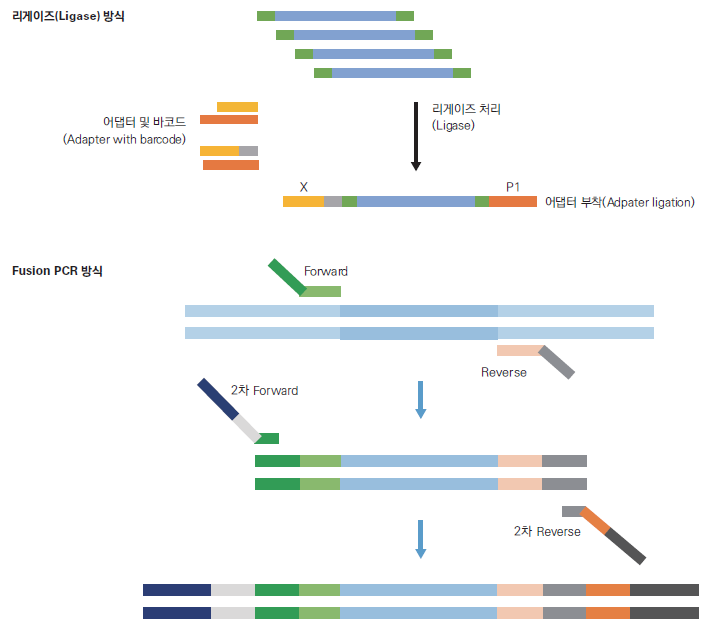
| 2 | 프로브 교합(Probe hybridization) 방식
프로브 교합(Probe hybridization) 방식은 보통 80~120 bp 정도 길이의 타겟 유전자에 특이적인 올리고뉴클레오티드 프로브를 고안하여 샘플 DNA 라이브러리에 교합(hybridization)시키는 방식이다. 프로브 끝에는 바이오틴(biotin)이 붙어 있어 아비딘(avidin)이 붙은 자성 비드(magnetic bead)에 결합 가능하도록 고안되어 있다. 따라서 프로브와 프로브에 결합된 DNA 라이브러리는 자성 비드에 함께 붙게 되고, 이것을 자석을 이용하여 선택적으로 걸러내게 된다. 라이브러리에 포함된 어댑터에 의한 비특이적인 교합을 방지하기 위해 블로킹 올리고(blocking oligonucleotide)를 첨가하기도 한다.
프로브교합 방식은 PCR 앰플리콘 방식에 비해 더 많은 양의 DNA가 필요하고 검사에 소요되는 시간이 길다는 단점이 있으나, 프로브 사이의 간섭이 덜하여 많은 수의 타겟을 비교적 고르게 선별할 수 있는 장점이 있다. 따라서 검사해야 할 타겟이 많은 경우나 전체 유전자를 검사하는 엑솜(exome)과 같은 경우 사용되는 방식이다. 또한 프로브교합 방식은 염기서열 변화뿐 아니라 유전자 복제 변이 수(copy number variation) 검출에도 분석이 가능한 장점이 있다.

5. NGS 분석 알고리즘
| 1 | NGS 데이터 정도관리 - quality control, QC
1) NGS 데이터 정도관리의 필요성
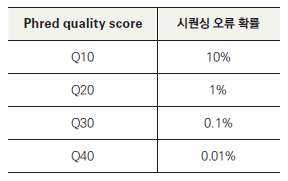
NGS는 기술적 한계와 실험적 원인에 의한 다양한 오류(error)의 가능성이 있다. NGS 염기서열분석 결과의 원데이터(raw data)에서는 추정 오류 확률을 수치로 나타내며 Phred 점수가 각 염기의 품질을 나타내는 지표로 활용된다(수식 1). Phred 점수 20점은 해당 염기서열 결과가 오류일 확률이 1%이라는 것을 의미하며, 30점은 0.1%의 오류 확률을 의미한다(표 5). 일반적으로 Q30 이상의 Phred 점수를 보이는 염기는 시퀀싱 품질이 우수하다고 판단하여 분석에 활용된다. 각 시퀀싱 리드(read)의 염기서열과 Phred 점수를 같이 표시한 것을 FASTQ 파일이라 부른다.
QPhred = -10 log10 P(error)
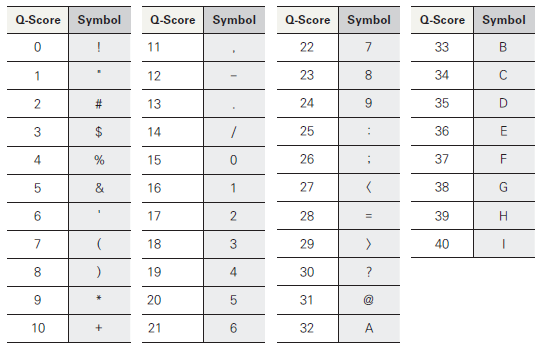
FASTQ 파일은 시퀀스 고유ID, 각 리드의 시퀀스, “+” 기호, 시퀀스의 각 베이스 별 Phred 점수로 된 네 개의 줄로 구성되어 있다. Phred 점수는 ASCII 심볼로 표시한다.

출처: 식품의약품안전처 식품의약품안전평가원 - 차세대염기서열분석 해설서
'[분자생물학] NGS' 카테고리의 다른 글
| NGS(Next Generation Sequencing) 기반 유전자 검사의 이해 - 심화용 (6) (0) | 2022.08.09 |
|---|---|
| NGS(Next Generation Sequencing) 기반 유전자 검사의 이해 - 심화용 (5) (0) | 2022.08.09 |
| NGS(Next Generation Sequencing) 기반 유전자 검사의 이해 - 심화용 (3) (0) | 2022.08.09 |
| NGS(Next Generation Sequencing) 기반 유전자 검사의 이해 - 심화용 (2) (0) | 2022.08.09 |
| NGS(Next Generation Sequencing) 기반 유전자 검사의 이해 - 심화용 (1) (0) | 2022.08.09 |



