| 7 | 유전체 데이터베이스 database 및 주석 annotation
NGS 변이가 검출되면 이 변이가 어떤 유전자의 어느 위치에 있는지, 데이터베이스에 등재되어 있는 변이 인지 등을 확인하여야 한다. 시퀀싱을 통하여 발굴하게 되는 변이의 수는 대략 수천 개에서 수백만 개에 이를 정도로 매우 많으므로 사람이 일일이 분석하기는 어렵고 각 변이마다 데이터베이스를 찾아주는 작업을 컴퓨터 알고리즘으로 자동화하는 것이 필요하며 이것을 주석(annotation)이라 부른다.
1) 정상인 데이터베이스 - Population database
질환과 관련이 없고 정상인에서도 발견될 수 있는 유전자 변이를 다형성(polymorphism)이라 부른다. NGS 검사를 하면 표준 염기서열과 다른 많은 수의 변이가 발견이 되고 대부분은 다형성 변이이다. 따라서 다형성 변이를 걸러내는 작업이 필요하고 여기에는 정상인 데이터베이스에서 발견되는 변이의 빈도(population frequency)가 중요하다. 이러한 데이터베이스는 특별한 건강의 문제 없이 생활하고 있는 정상 성인을 모아 전장 엑솜(whole exome) 검사를 하여 변이의 빈도를 측정한다. 특정 질환의 환자를 대상으로 변이 정보를 수집하지 않는다는 점에서 정상인 변이 데이터베이스라고 편의상 정의할 수 있지만, 아 직 발병하지 않은 일부 환자 혹은 돌연변이는 가지고 있지만 질환에 이환되지 않은 일부 보인자(carrier)의 변이가 포함될 수 있을 가능성이 있다. 그러한 변이의 빈도는 상대적으로 낮을 것으로 예상되므로 정상인 데이터베이스는 많은 질환 유전체 연구에서 연구자가 발굴한 변이의 빈도가 얼마나 되는지 파악하고 비교하는 용도로 널리 활용된다.

2) 질환 돌연변이 데이터베이스 - Mutation database
질환에 이환된 환자에서 유전검사를 하여 논문에 보고하거나 임상 검사 리포트를 한 것을 모아 놓은 데이터베이스로서 유전성질환과 관련된 생식세포 돌연변이 위주로 구성된 데이터베이스(ClinVar, HGMD, LOVD 등)와 암 돌연변이 위주의 데이터베이스(COSMIC, TCGA, ICGC 등)가 있다. 대부분 무료로 제공되나 HGMD 처럼 유료 데이터베이스도 있다.

3) 변이 주석 annotation 프로그램
변이에 주석을 달아주는 데에는 SNPeff, annovar, variant effect predictor(VEP) 등의 프로그램이 대표적으로 활용된다. 이러한 프로그램들은 데이터베이스의 해당 변이를 찾아 줄 뿐 아니라, DNA 변화가 생겼을 때 예측되는 아미노산 변화까지 계산해 준다. DNA 변화로 인해서 번역(translation) 과정의 조기 종료(premature termination)가 되는 nonsense 혹은 frameshift 변이의 경우 단백질의 기능 상실(loss-of-function)을 예측할 수 있다. 반면 단백질 아미노산이 일부만 변하는 missense 변이의 경우 그 해석이 어려운 경우가 많으며 단백질 구조에 미치는 영향을 예측하는 여러 in silico 알고리즘의 결과를 주석 프로그램에서는 제공해 준다.

| 8 | 컴퓨터 시뮬레이션 in silico 알고리즘
염기서열의 변화가 단백질의 구조나 기능에 어떤 영향을 줄 것인지를 예측하기 위해 컴퓨터 시뮬레이션(in silico) 알고리즘을 사용한다. 단백 변화(Misssense) 변이의 예측 알고리즘은 주로 진화 과정에서의 보존성(evolutionary conservation)을 점수화 하여 예측을 한다. 예를 들어, 어떤 단백질의 특정 위치의 아미노산이 진화 과정을 거치는 동안 변하지 않았다면 보존되었다(conserved) 판단하고 해당 위치의 아미노산은 중요한 역할을 하는 것으로 가정하여 아미노산이 바뀔 경우 병원성을 가질 가능성이 높아질 것으로 예측하는 방식이다. 이 이외에 단백질의 성상, 구조 등을 계산식에 포함하는 등 여러 알고리즘에서 다양한 방식을 사용하기 때문에 알고리즘마다 결과가 다르게 도출된다. RNA 스플라이스 위치(splice site) 변이 예측은 스프라이싱과 관련된 염기서열 모티프(motif) 및 엔트로피(entropy) 등을 분석하는 방식의 알고리즘들이 많다.
현재 수 많은 컴퓨터 시뮬레이션 알고리즘들이 개발되어 있으며 각각의 정확도와 특이도는 중등도(대략 60~80%) 정도 된다. 대부분의 알고리즘들이 병적인 변이로 과하게 치우쳐 예측하는 경향(overprediction)을 보이기 때문에 이 예측만을 가지고 변이의 분류에 사용하거나 임상적 결정을 내리는 것은 지양해야 한다. 이러한 문제 때문에 미국의학유전학회(American College of Medical Genetics and Genomics, ACMG) 가이드라인에서는 여러 개의 알고리즘을 동시에 사용하여 분석한 후 결과가 모두 일치할 경우만 참고를 하고 이 또한 보조적인(supporting) 근거로만 활용하도록 권고하고 있다.


| 9 | 데이터 시각화 - visualization
NGS 데이터의 오류 여부와 데이터 구조, 주변의 염기서열 및 데이터베이스 주석 여부 등을 시각화하여 눈으로 직접 확인이 가능하게 해주는 프로그램들이 있으며, 미국 Broad 연구소에서 개발한 Integrated Genome Viewer(IGV) 프로그램이 많이 사용된다.
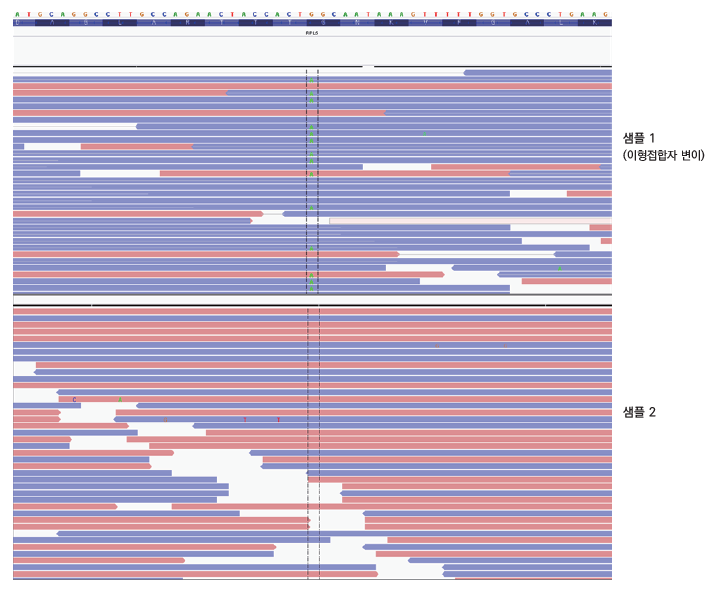
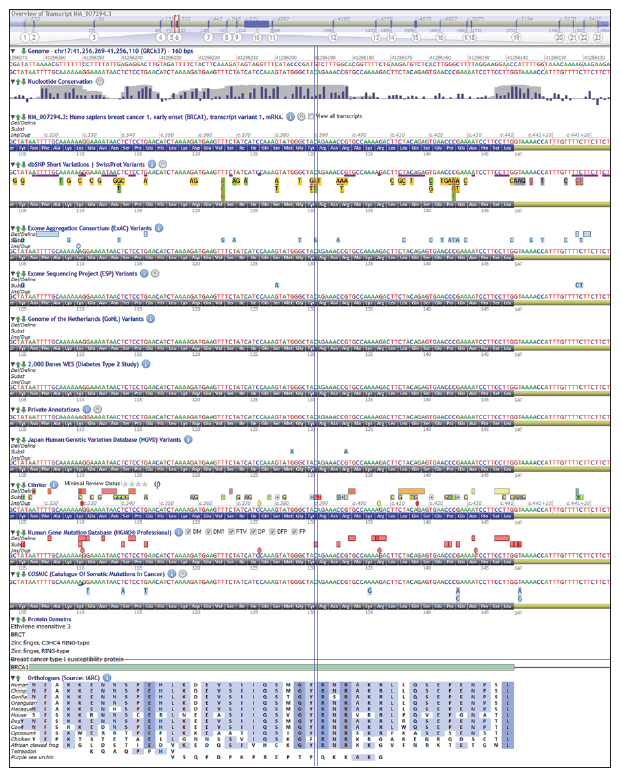
출처: 식품의약품안전처 식품의약품안전평가원 - 차세대염기서열분석 해설서
'[분자생물학] NGS' 카테고리의 다른 글
| NGS(Next Generation Sequencing) 기반 유전자 검사의 이해 - 심화용 (10) (0) | 2022.08.10 |
|---|---|
| NGS(Next Generation Sequencing) 기반 유전자 검사의 이해 - 심화용 (9) (0) | 2022.08.10 |
| NGS(Next Generation Sequencing) 기반 유전자 검사의 이해 - 심화용 (7) (0) | 2022.08.09 |
| NGS(Next Generation Sequencing) 기반 유전자 검사의 이해 - 심화용 (6) (0) | 2022.08.09 |
| NGS(Next Generation Sequencing) 기반 유전자 검사의 이해 - 심화용 (5) (0) | 2022.08.09 |



